Lundi 4 mars : Modélisation et énergies marines … et autres
Début à 14h30
Yann-Hervé De Roeck
Titre : Dans le domaine applicatif du développement des énergies marines renouvelables, quelques exemples d'interaction fine entre biologie et mathématiques
Yvon Maday
Titre : A propos des bases réduites
Francis Mairet
Titre : Metabolic modelling of microalgae during nitrogen starvation
Résumé : Microalgae are promising microorganisms for cosmetics, pharmaceuticals, feed, energy… However, in many fields, production yields are yet to be improved to obtain a sustainable and cost-effective process. This goal can be achieved thanks to the powerful tools of mathematical modeling and optimization, which can help to understand the intracellular mechanisms at place. Recently, a dynamic metabolic modeling framework – called DRUM – has been proposed in order to handle non-balanced growth condition and hence accumulation of intracellular metabolites (Baroukh et al. 2014). In this framework, we propose a metabolic model including organic carbon excretion in order to consider the effect of nitrogen on growth and carbon storage. Our approach is validated with a high-throughput time series concerning the haptophyte Tisochrysis lutea submitted to nitrogen starvation and day/night cycles. This study highlights the need for metabolic modeling in dynamic conditions to better understand microalgal metabolism and potentially tune algae production, and it offers an alternative mechanistic explanation to the empirical Droop model.
Julien Salomon
Titre : Analyse de la Blade Element Momentum Theory, application à l'extraction d'énergie fluviale Cliquer ici pour voir le diaporama de l'exposé (0.8 Mo)
Résumé : La Blade Element Momentum Theory fournit un modèle qui permet d'évaluer numériquement l'efficacité d'une hélice. L'avantage de l'algorithme associé réside dans la décomposition du calcul en deux parties : un modèle 2D qui rapporte les forces de portance et de traînée associées au profil considéré, et un système réduit d'équations scalaires qui décrit les forces macroscopiques appliquées sur l'hélice. Dans cet exposé, nous présenterons les hypothèses nécessaires pour obtenir l'existence de solution(s) de ce dernier système. De plus, nous prouvons la convergence d'un algorithme à point fixe qui peut être utilisé pour le résoudre numériquement. Nous terminons cet exposé avec quelques résultats obtenus en appliquant ce modèle à un extracteur de courant de rivière flottant.
Mardi 5 mars : Données et modélisation … et autres
Vincent Calvez
Titre : Equilibres dans des modèles de génétique quantitative Cliquer ici pour voir le diaporama de l'exposé (4.1 Mo)
Jean Yves Chemin
Titre : LJLL et fluides géophysiques: quelques souvenirs personnels
Philippe Ciarlet
Titre : Variations sur un lemme de J.-L. Lions Cliquer ici pour voir le diaporama de l'exposé (1.2 Mo)
Résumé : Il est bien connu qu'un certain lemme dû à J.-L. Lions, qu'on appelera ici « le » lemme de J.-L. Lions, joue un rôle fondamental dans la théorie de certaines équations aux dérivées partielles elliptiques. Mais ses démonstrations connues dans le cas des domaines à frontière seulement lipschitzienne sont difficiles.
Dans cet exposé, on montrera que le lemme de J.-L. Lions est en fait équivalent à un certain nombre d'autres résultats fondamentaux, par exemple une inégalité due à J. Necas, ou à la surjectivité de l'opérateur divergence entre des espaces fonctionnels appropriés. Certains de ces résultats équivalents ont des démonstrations « directes », par exemple la preuve constructive de la surjectivité de l'opérateur divergence due à M.E. Bogovskii, qui repose entre autres sur la théorie des intégrales singulières de Calderon-Zygmund. Toute preuve "directe" de chacun de ces résultats fournit donc une preuve du lemme de J.-L. Lions.
Patrick Gallinari
Titre : Physico-statistical systems – a new challenge for machine learning Cliquer ici pour voir le diaporama de l'exposé (2.8 Mo)
Résumé : There are two main paradigms today for modeling complex natural or artificial phenomena. The physical one mainly developed since the 19th century, relies on the analytical description of the scientist prior knowledge of an underlying process. This prior knowledge has been accumulated over decades by generations of scientists analyzing and formalizing the laws of nature. This is today the main framework used in physics. With the availability of large amounts of data gathered from different types of sensors, this classical way of thinking is challenged by the more recent statistical Machine Learning paradigm which offers a prior agnostic approach based on the pure exploitation of data. The convergence of these two approaches is certainly one of the great scientific challenges for the next decades. In this presentation we introduce some preliminary work exploring this direction focusing on the modeling of spatio-temporal processes, typically problems from fluid dynamics. We present two use cases, one is an example of how to incorporate prior physical knowledge in a deep learning system, and the second one shows how to learn dynamical systems obeying general evolution equations using a deep learning approach.
Hervé Le Dret
Titre : Données et mécanique des structures Cliquer ici pour voir le diaporama de l'exposé (1.2 Mo)
Résumé : On n’y échappe pas : la donnée s’invite désormais en calcul des structures avec la «data driven computational mechanics ». Alors, véritable changement de paradigme ou miroir aux alouettes ? On tentera d’y voir un peu plus clair à partir notamment des travaux récents de Kirchdoerfer-Ortiz et Conti-Müller-Ortiz. Ces derniers ont dans la foulée mis en évidence d’un point de vue plus théorique une nouvelle notion de relaxation qui mérite sans doute que l’on s’y arrête.
Marie Postel
Titre : La modélisation mathématique de la neurogénèse dans le cortex cérébral Cliquer ici pour voir le diaporama de l'exposé (5 Mo)
Résumé : La neurogénèse corticale est un processus combinant les divisions de cellules progénitrices de deux principaux types. Les effectifs, taux de divisions et durées de cycles de ces progéniteurs conditionnent l'effectif final de neurones. Pour comprendre les rôles respectifs de ces facteurs dans la neurogenèse, nous combinons des études expérimentales in vivo avec une modélisation mathématique et une simulation numérique de la dynamique des progéniteurs. Un formalisme multi échelles nous permet de suivre la progression de types de progéniteurs spécifiques tout au long des phases du cycle cellulaire. Nous ajustons les valeurs numériques du modèle de manière à correspondre avec des effectifs cellulaires expérimentaux obtenus sur un modèle de souris.Des résultats expérimentaux obtenus sur des sujets présentant une mutation ont également pu être décrits par le modèle, et nous avons analysé les différences en termes de cinétique cellulaire et du nombre final de cellules.
Karim Ramdani
Titre : Edition scientifique : un rapide survol des évolutions en cours Cliquer ici pour voir le diaporama de l'exposé (3.6 Mo)
Résumé : Durant cet exposé, je présenterai les différents modèles économiques en vigueur dans l’édition scientifique et dresserai un panorama des évolutions en cours. Je m’intéresserai plus particulièrement à l’émergence du modèle auteur-payeur (parfois désigné par le terme Gold Open Access) et je tenterai de montrer les risques qu’il présente. Enfin, je conclurai en évoquant quelques pistes de solutions possibles à l’échelle individuelle ou institutionnelle.
Mercredi 6 Mars : Modélisation et métabolisme… et autres
Jean Clairambault
Titre : Cancer as a default of coherence between tissues in metazoans: what mathematical models should be developed to help prediction, prevention and treatment of cancer, avoiding drug resistance? Cliquer ici pour voir le diaporama de l'exposé (13.4 Mo)
Albert Cohen
Titre : Réduction de modèle et données ponctuelles
Résumé : Les méthodes de réduction de modèle telles que les bases réduites ont des propriétés d’approximation supérieures à celles des approches classiques (éléments finis, splines, Fourier, ondelettes) pour certaines familles de solutions d’équations aux dérivées partielles paramètrées. Lorsque la valeur du paramètre est inconnue et que l’on observe la solution en plusieurs points, il est ainsi naturel de chercher à en reconstruire une version approchée dans ce type d’espaces. Nous discuterons deux méthodes (interpolation et moindres carrés) qui conduisent dans chaque cas à rechercher la distribution des points d’observation permettant une reconstruction optimale du point de vue de l’erreur obtenue.
Stéphane Egée
Titre : Red Blood Cell Cationic conductances in Health and Disease Cliquer ici pour voir le diaporama de l'exposé (1.6 Mo)
François Fages
Titre : Calculs analogiques dans les programmes biochimiques naturels et synthétiques Cliquer ici pour voir le diaporama de l'exposé (21.5 Mo)
Résumé : Lorsqu’un informaticien cherche à comprendre comment la cellule effectue les calculs nécessaires à son maintien dans l'environnement, au traitement des signaux qu’elle reçoit, à la prise de décision, et aux commandes de divers processus comme par exemple le cycle de division cellulaire, la nature continue des processus d’interactions protéiques, qui sont omniprésents dans la cellule, nous oblige à envisager des modèles de calcul analogique.
Cela est en contradiction avec la principale leçon de l’informatique dont les 80 ans d’histoire ont montré que dans le domaine électronique, seul le calcul digital était capable de passer à l’échelle des grands systèmes.
La preuve récente de Turing-complétude des réseaux de réactions chimiques (CRNs) continus, s’appuyant sur des résultats également récents de calculabilité et complexité analogiques, a permis de réconcilier sur le plan théorique les deux mondes, en montrant que toute fonction calculable sur les réels (i.e. par une machine de Turing en précision arbitraire passée en argument) peut-être calculée par un système d’équations différentielles polynomiales (par approximation du résultat sur une composante du système), et en fait par un CRN élémentaire sur un nombre fini d’espèces moléculaires.
Dans cette vue du calcul biochimique, les concentrations continues des espèces moléculaires (protéines en grand nombre) constituent le support de l’information; les programmes sont décrits formellement par l’ensemble des réactions chimiques possibles entre les protéines exprimées du génome ainsi qu'avec les molécules de l’environnement; les changements d'activation des gènes (de nature digitale) déterminent les changements de programmes et constituent en quelque sorte des metaprogrammes.
De la preuve du résultat théorique, nous avons tiré un compilateur de toute fonction réelle calculable (présentée comme solution d'équations différentielles polynomiales), ou de programmes mixtes analogiques-digitaux, en CRNs finis. Nous pouvons utiliser ce compilateur pour concevoir des CRNs artificiels en biologie synthétique ou effectuer des comparaisons avec les circuits naturels en biologie des systèmes.
Nos résultats expérimentaux ont porté sur l’implantation biochimique des fonctions trigonométriques (oscillateurs comparables par exemple aux circuits naturels d’horloge circadienne), des fonctions sigmoïdes (interrupteurs et convertisseurs analogiques-digitaux comparables aux circuits naturels de signalisation comme MAPK), des portes logiques (conception de vésicules artificiels de diagnostic différentiel des diabètes), de la séquentialité et des boucles (programmes impératifs comparables aux circuits naturels de commande du cycle de division cellulaire).
Ce cadre d’étude nous amène aussi à développer des méthodes alternatives de conception approchée de CRNs par évolution artificielle avec l’espoir de les comparer plus facilement aux circuits naturels.
Camilla Fiorini
Titre : Sensitivity analysis for nonlinear hyperbolic systems of conservation laws Cliquer ici pour voir le diaporama de l'exposé (7.8 Mo)
Antoine Gloria
Titre : 50 ans d'homogénéisation au LJLL Cliquer ici pour voir le diaporama de l'exposé (6.8 Mo)
Résumé : Dans cet exposé, je présenterai une sélection de résultats désormais classiques de la théorie de l'homogénéisation périodique démontrés au Laboratoire Jacques-Louis Lions depuis sa création, tant du point de vue de l'analyse des EDP que de la modélisation et la simulation. Je conclurai par des perspectives dans le cadre de l'homogénéisation stochastique.
François Murat
Titre : Comment la compacité par compensation a été inventée par Luc Tartar
Anne Siegel
Titre : Raisonner sur des réseaux biologiques à grande échelle avec des données hétérogènes Cliquer ici pour voir le diaporama de l'exposé (14.8 Mo)
Résumé : Le domaine des sciences de la vie est attendu pour produire dans la prochaine décennie des données aux caractéristiques particulièrement complexes: elles seront non seulement massives, mais aussi multi-échelles, incomplètes, hétérogènes et surtout interdépendantes. Dans ce contexte, les approches de modélisation basées sur des systèmes dynamiques sont difficilement applicables à l'échelle de la biologie cellulaire et moléculaire, puisque les systèmes ne sont pas identifiables. Pour dépasser ce verrou, nous présenterons une stratégie pour étudier le métabolisme des systèmes biologiques à grande échelle. Cette stratégie s'appuie sur des approches de représentation des connaissances, qui permettent de modéliser les interactions dans un système et de sur-approximer les conséquences de la dynamique du système. On en déduit des propriétés du système biologique en résolvant des problèmes d'optimisation combinatoire. Nous illustrerons cette approche par l'exemple des interactions entre un hôte et son microbiome, étudiées dans le cadre d'une collaboration de long terme avec la station biologique de Roscoff.
Michele Thieullen
Titre : Modélisation de l’activité électrique d’une cellule excitable par les processus de Markov déterministes par morçeaux (PDMP)
Résumé : Une cellule excitable (un neurone, le coeur, un muscle) est capable de répondre à certaines stimulations électriques par un potentiel d’action grâce à la présence de canaux ioniques au niveau de sa membrane. Certains canaux ioniques sont spécifiques à un certain type d’ions (sodium, potassium, calcium par exemple) et influent sur la permitivité de la membrane. La modélisation déterministe repose sur des modèles à conductances, dont les plus connus sont celui d’Hodgkin et Huxley et celui de Morris et Lecar. Les modèles à conductances sont dits biologiquement réalistes car ils contiennent des variables qu’on peut associer aux canaux ioniques, par opposition par exemple au modèle de FitzHugh-Nagumo. Mais les modèles déterministes décrivent un neurone qui aurait une infinité de canaux ioniques. Dans l’exposé on présentera la modélisation stochastique par les processus de Markov déterministes par morçeaux (PDMP) qui correspondent au cas où le nombre de canaux est fini. Pour ces modèles on décrira des résultats de nature théorique ainsi que des résultats concernant leur simulation.
Alain Viari
Titre : Génomique des cancers: de la recherche à la clinique Cliquer ici pour voir le diaporama de l'exposé (17.6 Mo)
Résumé : L'analyse bioinformatique des génomes tumoraux s'est considérablementdéveloppée durant la dernière décennie dans le domaine de la rechercheen cancérologie. Elle permet d'obtenir des informations très précisessur les altérations génomiques mais aussi sur certains mécanismes moléculairesà l'oeuvre dans la cancérogénèse. J'en donnerai quelques exemples dans lecas des cancers du sein dits « amplifiés sur HER2 » et de certains carcino-sarcomes.Plus récemment, un glissement s'opère de la recherche fondamentale vers larecherche clinique, voire le diagnostic. En France, ce mouvement s'est amplifié avec le Plan France Médecine Génomique 2025. Sortir du cadre de la recherchestricto-sensu soulève de nouvelles questions d'ordre organisationnel mais aussi éthique et sociétal qu'il me semble également intéressant d'évoquer.
Jeudi 7 mars : Modélisation en écologie… et autres
Sakina Ayata
Titre : Modélisation mécanique et statistique en écologie marine : exemples pour l'étude du plancton marin Cliquer ici pour voir le diaporama de l'exposé (13.2 Mo)
Résumé : Dans les océans, le plancton regroupe une grande diversité d'organismes flottants au grè des courant (comme par exemple les microalgues, de petits crustacés comme le krill, des organismes gélatineux comme les méduses, ou encore des larves d'invertébrés ou de poissons). Le plancton joue un rôle clef dans le fonctionnement des écosystèmes marins dans les cycles biogéochimiques globaux. En effet, il est responsable de la production de près de la moitié de l'oxygène que nous respirons et joue un rôle central dans le cycle du carbone, à travers l'export de carbone vers le fond des océans, et contribue ainsi à la régulation du climat de notre planète. Enfin, il est à la base des réseaux trophiques marins qui alimentent les pêcheries et donc les populations humaines. Etant donnée la difficulté d'observer le plancton in situ, l'utilisation de modèles numériques est un outil puissant pour l'étudier et tester des hypothèses pour expliquer la structure et le fonctionnement des écosystèmes planctoniques.
Après avoir présenté les grands enjeux de la recherche actuelle en écologie planctonique, j'illustrerai, à travers plusieurs exemples, comment les modèles mathématiques sont utilisés pour étudier le plancton marin. Il sera en particulier question de modèles eulériens et lagrangiens de dispersion, de modèles méchanistiques de flux de matière, et de modèles statistiques de distribution d'espèces.
Simon Dittami
Titre : Response of algal holobionts towards environmental changes Cliquer ici pour voir le diaporama de l'exposé (10.7 Mo)
Michel Duprez
Titre : Problèmes de contrôle liés aux mouvements de foules Cliquer ici pour voir le diaporama de l'exposé (8.1 Mo)
Roland Glowinski
Titre : On the numerical solution of nonlinear eigenvalue problems for the Monge-Ampère operator: an operator-splitting approach Cliquer ici pour voir le diaporama de l'exposé (1.5 Mo)
Résumé :
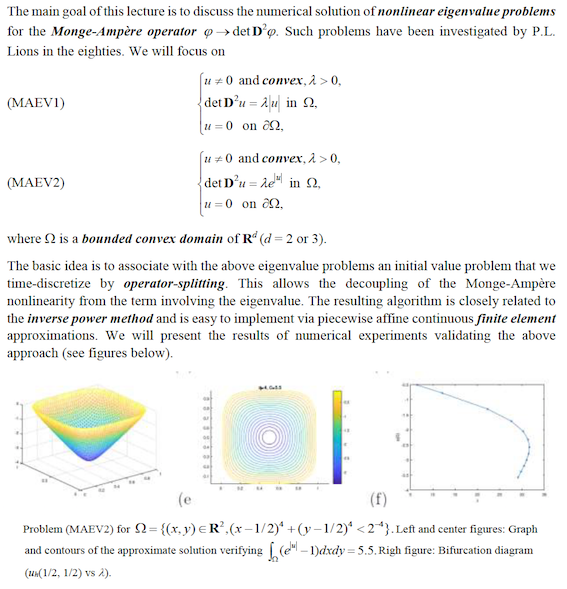
Fabrice Not
Titre : Adaptation et diversité en milieu marin Cliquer ici pour voir le diaporama de l'exposé (6.7 Mo)
Olivier Pironneau
Titre : Mathematics, Supercomputing and Multidisciplinarity Cliquer ici pour voir le diaporama de l'exposé (4.2 Mo)
Bastien Polizzi
Titre : Mixture models for phototrophic biofilms and gut microbiota ecology Cliquer ici pour voir le diaporama de l'exposé (3.4 Mo)
Résumé : The framework of mixture theory allows describing complex systems of heterogeneous fluids at the mesoscale which is an intermediary scale between micro and macro. This formalism generalises Euler equations and uses partial differential equations to model multicomponent fluids. Therefore, mixture theory is particularly well adapted to describe complex biological ecosystems such as photosynthetic microalgae biofilm and gut microbiota ecology.
The purpose of the talk will be to introduce mixture theory formalism and then present these two applications. In both cases (biofilm and gut ecology), the objective is to quantify the influence of mechanical, ecological and chemical mechanisms considered in the models. For each model the presentation structured as follow. First, the context will be present. Then the model will be described. Eventually, the numerical schemes and simulations results will be commented.
Philippe Potin
Titre : IDEALG : un projet d’investissements d’avenir qui a permis dix ans de coopération entre les mathématiques, l’informatique et la biologie des macro-algues et de leurs microbiotes Cliquer ici pour voir le diaporama de l'exposé (14.4 Mo)
Résumé : Coordonné par la Station Biologique de Roscoff, le projet IDEALG a pour objectif de consolider et accroître les connaissances nécessaires pour développer la filière des grandes algues marines en France. IDEALG repose sur trois axes d'études complémentaires qui permettent d'aborder le développement du secteur en intégrant à la fois les moteurs et les goulets d'étranglement potentiels au niveau de la recherche fondamentale ainsi que de la recherche appliquée et du transfert de connaissances et de technologies. L’axe 1 Génomique et recherche post-génomique vise à fournir les données nécessaires pour le potentiel des algues en termes de diversité génétique, de voies métaboliques et d'interactions avec d'autres organismes ou leur environnement. L’axe 2 a conduit au développement d'outils analytiques et biotechnologiques et d'études chimiques pour explorer puis contrôler le métabolisme des algues à des fins industrielles. Enfin l’axe 3 développe la culture des algues et la conservation des ressources génétiques pour se diriger vers la domestication de plusieurs espèces. IDEALG entame sa 9ième année avec un nombre impressionnant de réalisations. En particulier le développement d’outils génétiques, chimiques et mathématiques dans le but de mieux comprendre la physiologie, le métabolisme et l’évolution des algues dans leur environnement.
Nastassia Pouradier Duteil
Titre : Control and Pattern Formation in Collective Dynamics Cliquer ici pour voir le diaporama de l'exposé (3.1 Mo)
Denis Roze
Titre : Adaptation locale et évolution de la reproduction sexuée Cliquer ici pour voir le diaporama de l'exposé (0.9 Mo)
Résumé : La génétique des populations s’intéresse aux différents facteurs agissant sur les fréquence des allèles au sein des populations. Au cours de cet exposé je présenterai plusieurs modèles de populations situées le long d’un habitat linéaire, et entre lesquelles les individus peuvent se déplacer par migration. Les effets de la sélection (favorisant certaines combinaisons génétiques dans certaines populations) et de la migration sur les fréquences des allèles peuvent être représentées par des équations de diffusion. Différentes combinaisons d’allèles peuvent être maintenues dans différentes populations lorsque ces combinaisons sont incompatibles (produisant des individus hybrides maladaptés) ou lorsqu’elles sont impliquées dans des adaptations locales. Je présenterai différentes questions en cours d’études, relatives à la dynamique de tels systèmes et à leur effet sur l’évolution du mode de reproduction des organismes.
Benoît Sarels
Titre : Comportement collectif de plusieurs fronts d’avance en génétique des populations : agrégation et accélération induite Cliquer ici pour voir le diaporama de l'exposé (18 Mo)
Résumé : La propagation d'un gène avantageux au sein d'une population peut être décrite par une équation qui possède des solutions sous forme de fronts d'avance. Dans le cas de plusieurs loci, le phénomène de recombinaison génétique introduit un couplage entre les différents allèles, donc une interaction entre les fronts.
Nous présenterons ici la construction du système d'équations décrivant le cas de deux loci à deux allèles. Nous montrerons l'intérêt qu'il y a à étudier ce système dans les repères mobiles attachés aux fronts pour mettre en évidence des phénomènes tels que l'agrégation de fronts et l'accélération induite par l'agrégation.
Enfin, l'extension à un nombre arbitraire de loci donnera une jolie construction d'un système "à la Kuramoto" pour lequel nous présenterons des questions et développements récents.
Vendredi 8 mars : Modélisation en SHS… et autres
Yves Achdou
Titre : Some aspects of mean field games Cliquer ici pour voir le diaporama de l'exposé (1.4 Mo)
Résumé : Recently, an important research activity on mean field games (MFGs for short) has been initiated by the pioneering works of Lasry and Lions: it aims at studying the asymptotic behavior of stochastic differential games (Nash equilibria) as the number n of agents tends to infinity. The field is now rapidly growing in several directions, including stochastic optimal control, analysis of PDEs, calculus of variations, numerical analysis and computing, and the potential applications to economics and social sciences are numerous.
In the limit when n tends to infinity, a given agent feels the presence of the others through the statistical distribution of the states. Assuming that the perturbations of a single agent's strategy does not influence the statistical states distribution, the latter acts as a parameter in the control problem to be solved by each agent. When the dynamics of the agents are independent stochastic processes, MFGs naturally lead to a coupled system of two partial differential equations (PDEs for short), a forward Fokker-Planck equation and a backward Hamilton-Jacobi-Bellman equation. When there is a common noise, the situation becomes more complicated: one has to see the value function of the game as a function of the distribution of states; when the state space is not finite, this means solving a PDE in infinite dimension.
Even in the former simpler case, the system of PDEs has closed form solutions in very few cases only. Therefore, numerical simulation are crucial in order to address applications. The present mini-course will be devoted to numerical methods that can be used to approximate the systems of PDEs.
Several aspects of mean field games related to modelling and numerical simulations will be addressed.
Matthieu AlfaroTitre: Modèles replicator-mutator en biologie évolutive Cliquer ici pour voir le diaporama de l'exposé (0.8 Mo)Résumé : Depuis Kimura et les années 60, les équations dites replicator-mutator sont des modèles centraux en biologie évolutive. Je présenterai des résultats mathématiques très récents sur ces modèles (concernant des fitness non majorées ou localisées) qui éclairent sur le phénomène de branchement et, le cas échéant, le nombre de phénotypes sélectionnés. Si le temps le permet, je discuterai l'introduction d'un processus de naissance et mort dans le modèle.
Laurent Boudin
Titre : Recent mathematical and modelling developments about the Hegselmann-Krause model
Résumé : In an ongoing work with Francesco Salvarani (Université Paris Dauphine & Università di Pavia, Italy) and Emmanuel Trélat (Sorbonne Université), we provide a new mathematical insight on the Hegselmann-Krause model in the nonsymmetric case. We investigate an original form of the variance to obtain the convergence towards equilibrium and discuss the consequences on the model.
Fin de la conférence à 11h30
Excursion proposée le vendredi 8 mars après midi
Après la conférence, une excursion sur l'île de Batz est proposée aux participant(e)s ; elle consistera à
• prendre le bateau directement après la dernière conférence sans retourner à l'hôtel,
• déjeuner sur l'île (éventuellement formule sandwich),
• faire la ballade (*)
• revenir à l’hôtel "Le gulf stream" du CNRS pour récupérer les bagages (que chacun(e) aura préparé le matin),
• prendre un taxi à plusieurs pour aller prendre le train de 17h44 à Morlaix,
• arriver à Paris Montparnasse à 21h04.
(*) Une visite du Jardin exotique Georges Delaselle était prévue dans la ballade, mais le jardin sera encore fermé début mars ; il y aura néanmoins plein de belles choses à voir, en particulier le phare avec un panorama qui va des Sept-Îles à l'est jusqu'à l'Ile-Vierge à l'ouest, en passant par l'entrée de la Baie de Morlaix et la silhouette des Monts d'Arrée qui se dessine à l'horizon sud.





